2. 北京城市学院 生物医药学部, 北京 100094
2. School of Biomedicine, Beijing City University, Beijing 100094, China
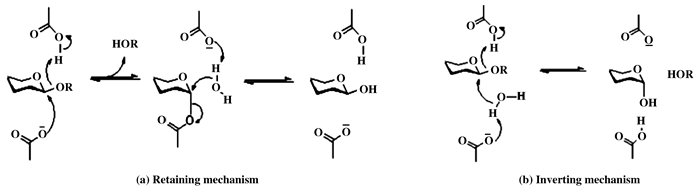
糖苷酶(Glycoside hydrolase, GH, EC3.2.1)是广泛存在于动物、植物、微生物体内的一类水解酶, 根据其水解底物的不同, 可分为淀粉酶(EC3.2.1.1)、纤维素酶(EC3.2.1.4)、木聚糖酶EC3.2.1.8)、β-葡萄糖苷酶(EC3.2.1.21)等195种酶.目前, 碳水化合物活性酶数据库(www.CAZy.org)根据氨基酸序列相似度大于30%的原则, 可将糖苷酶具体分为167个家族GH1-GH167;根据其催化功能的活性中心结构域的不同, 又可将这些家族归纳为GH-A—GH-R18个族.同一家族的糖苷酶由于具有相似的活性中心结构, 所以往往具有相同的催化机制, 根据催化反应时底物糖环构象的立体化学行为, 将糖苷酶的催化反应机制分为底物构象保持型和底物构象反转型[1-2](见图 1).
|
图 1 糖苷酶催化机制 Fig.1 Reaction mechanisms of glycosidases |
糖苷酶在工业、生物医药、食品等领域有着重要的应用价值, 如木质纤维素的生物降解、疾病治疗及糖苷化合物的生物催化转化[3-4].然而, 大多数天然糖苷酶的催化活性、稳定性及底物选择性难以满足实际应用要求, 需通过有效的酶工程策略对其结构和功能进行改造优化[5].传统的定向进化技术虽在酶分子改造领域取得了很多进展, 但它往往建立在多轮有益突变叠加的基础上, 存在突变体库大、筛选工作量大的问题[6].随着计算机技术和先进算法的不断开发与完善, 以及蛋白质序列、结构等生物信息的不断丰富, 计算机辅助酶分子设计已成为酶工程研究的重要手段[2, 7].计算机辅助酶设计结合了计算化学和生物信息学的方法, 通过理论计算便可获得传统实验技术难以得到的数据, 在蛋白质结构与功能、酶分子改造等研究领域发挥着越来越重要的作用[8-9].利用同源比对、分子对接以及动力学模拟等计算机辅助方法, 人们可以更深入地研究糖苷酶的结构特征、酶催化过程中的构象变化, 进而理解其催化机制和构效关系, 最终实现糖苷酶的定向设计与改造, 从而为促进其工业应用奠定基础.
1 计算机辅助生物信息同源比对随着Genbank数据库基因序列和蛋白质结构数据库PDB(Protein Data Bank)中结构信息的不断丰富, 同源比对成为研究糖苷酶结构与功能关系的有效策略.同源比对通常包括多序列比对、系统进化分析和结构比对分析.基于糖苷酶家族的系统进化分析, 对同源序列或结构进行比较, 可分析进化上的远近、鉴定保守性氨基酸及共同进化的重要氨基酸. 表 1总结了近年来同源比对应用于糖苷酶设计改造的部分报道.
| 表 1 基于同源比对的糖苷酶设计改造部分实例 Table 1 Some examples of glycosidase design and modification based on homology alignment |
多序列比对分析和系统进化分析方法已成为研究糖苷酶序列保守性和活性位点的标准工具. Pachl等[15]对GH5家族芦丁糖苷酶AnRut和其同源酶进行系统进化分析, 发现同源性较高的糖苷酶(见图 2, 红色编号), 进一步多序列比对确定了AnRut参与底物结合和发挥催化功能的关键活性位点(见图 3, 红色位点).

|
图 2 GH5家族芦丁糖苷酶AnRut的系统进化分析[14] Fig.2 Phylogenetic analysis of GH5 family AnRut |

|
图 3 AnRut与同源酶的多序列比对 Fig.3 Multiple sequence alignment of AnRut and homologous enzymes |
结构比对分析是酶结构分析的重要手段.通过对同源蛋白的三维结构进行叠合比较, 可分析引起同源蛋白功能差异的结构基础. Schiano等[16]对GH7家族超嗜热内切葡聚糖酶ReCel7B与同源结构的比对分析表明, ReCel7B结构表面的loop环刚度较高(见图 4, 蓝色结构), 且存在较多的糖基化位点(见图 4, 绿色位点), 从而揭示了其较高耐热机制的结构基础.

|
图 4 GH7内切葡聚糖酶ReCel7B(橘色)与同源酶TrCel7B(绿色)的结构叠合比对, 结构差异大的loop结构分别用蓝色和品红色表示 Fig.4 Stuctural comparison between GH7 endoglucanase ReCel7B (orange) and homologous endoglucanase TrCel7B (green), and loop structures with large structural differences are highlighted in blue and magenta for ReCel7B and TrCel7B, respectively |
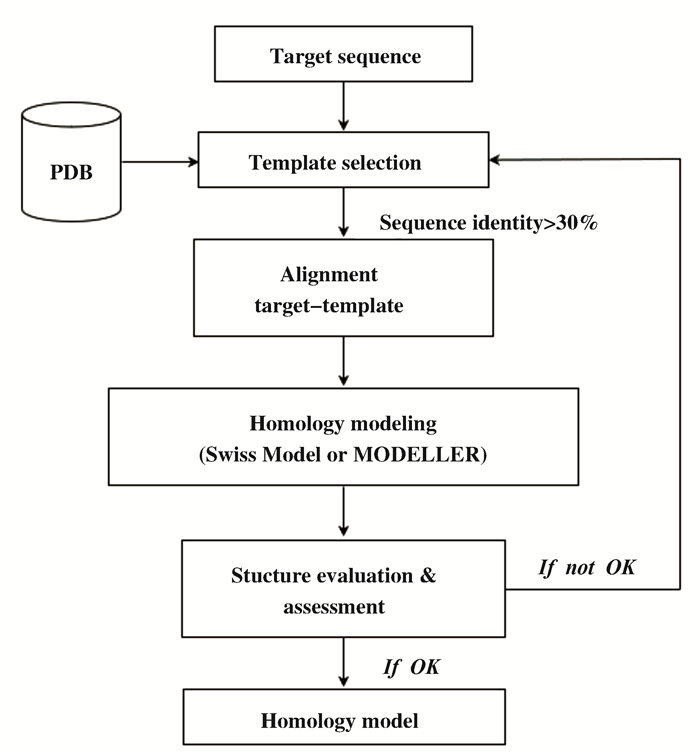
蛋白质结构解析是目前最有效的获取蛋白结构的技术手段, 但存在结构解析周期长、成本高等缺点[17].相比实验方法, 计算机辅助结构预测弥补了上述不足, 它利用已知目标氨基酸序列来构建空间结构模型.在研究尚未获得晶体结构的酶时, 大多数研究者以已解析的同源结构为模板, 模拟预测其三维结构模型, 进而在此基础上进行结构分析及分子设计[3, 5, 18].常用的结构预测方法主要有同源建模法、从头计算法和穿线法[19].其中, SWISS-MODEL同源建模已成为预测蛋白质结构的首选方法, 它只需从PDB蛋白质结构数据库找到与目标序列一致度大于30%的已知蛋白质结构的同源序列作为模板, 即可通过建模软件构建目标蛋白的结构模型.基于同源蛋白的结构建模流程可参见图 5.
|
图 5 SWISS-MODEL同源建模流程 Fig.5 Workflow of homology modeling using SWISS-MODEL |
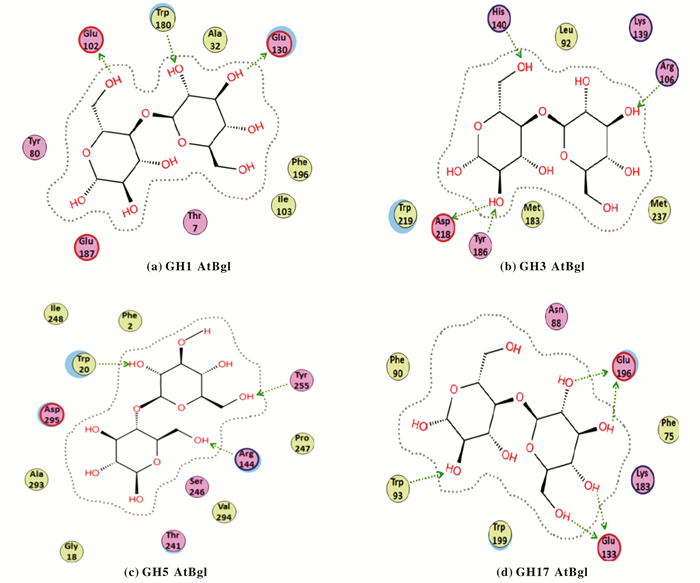
分子对接技术通过受体结构和配体结构间的几何互补、能量匹配来实现酶-底物复合物的对接.对于结构模拟获取的三维结构, 可利用分子对接软件进一步分析其结构特征、预测糖苷酶行使催化功能的关键位点. Dadheech等[20]通过同源建模、建立对接模型并进行酶与底物相互作用力分析, 揭示了4种家族的土曲霉来源的β-葡萄糖苷酶AtBgl参与底物纤维二糖水解的保守性氨基酸残基(见图 6), 为进一步设计具有高催化效率的β-葡萄糖苷酶提供了位点改造规律. Zhang等[21]对大肠杆菌β-葡萄糖醛酸苷酶进行分子对接, 进一步结构分析和定点突变, 发现突变后的559位丝氨酸(S)是酶催化过程中的关键残基, 突变体G559S相比野生酶活性提高2~6倍.
|
图 6 不同家族β-葡萄糖苷酶AtBgl与纤维二糖的分子对接[20] Fig.6 Molecular docking of different β-glucosidases with cellobiose[20] (绿色直线箭头代表相互作用力) (a) GH1 AtBgl; (b) GH3 AtBgl; (c) GH5 AtBgl; (d) GH17 AtBgl |
酶结构的构象变化对其行使催化功能是必要的.然而, 酶结构的柔性赋予酶催化机制的复杂性, 加大了人们认识酶催化过程中构象变化的难度.分子动力学模拟技术基于经典分子力学力场模拟生物大分子的原子运动, 可从不同角度指导酶设计改造.通过模拟酶在催化过程的构象变化, 不仅有助于分析决定酶催化功能的结构基础, 同时也促进了人们对糖苷酶催化机制的理解.
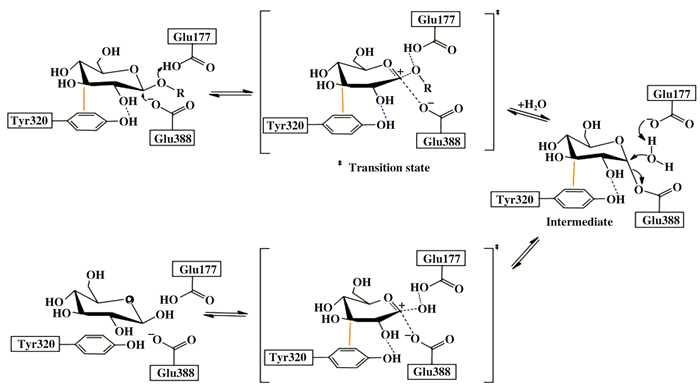
3.1 分子动力学模拟辅助分析糖苷酶催化机制分子动力学模拟技术用于预测决定酶催化分子机制的关键位点.来源于费氏新萨托菌Neosartorya fischeri的GH1家族β-葡萄糖苷酶NfBGL具有较高的催化活性, 在木质纤维素的生物转化、生物燃料的合成等方面展现出潜在的工业用途.为阐明决定该酶高催化活性的分子机制, Shanmugam等[22]基于该酶的同源结构, 对其进行了分子动力学模拟.随后通过模拟Tyr320突变体酶和野生酶与底物pNPG的相互作用差异, 发现底物结合口袋的Tyr320位点与底物pNPG的吡喃糖环之间存在氢键和π-σ相互作用(图 7).由此发现底物结合口袋的Tyr320是决定该酶高催化活性的关键位点.
|
图 7 分子动力学模拟分析Tyr320位点在酶催化过程中的作用机制[22] Fig.7 Molecular dynamics simulation analysis of the role of Tyr320 in the stabilization of the enzyme catalysis[21] (蓝色虚线和橘色实线分别代表酶与底物间的氢键和π-σ相互作用) (Blue dotted line: H-bonding, orange solide line: π-sigma interaction) |
工业酶通常需要具备较高的热稳定性、催化活性和底物特异性, 因此提高天然酶的催化性能对促进其工业应用具有重要意义.利用分子动力学模拟对加深认识酶结构与功能间的关系, 加快糖苷酶的设计改造进程发挥着重要作用.例如, 分子动力学模拟用于预测决定底物选择性的关键位点[23-25].来自哈茨木霉Trichoderma harzianum的GH7家族内切葡聚糖酶ThCel7B, 在木质纤维素的降解中发挥重要作用. Sonoda等[26]基于该酶的晶体结构解析, 利用分子动力学模拟揭示了酶分子与不同底物多糖的结合亚位点及其氢键相互作用网络(见图 8), 为该酶的选择性识别改造提供了理论依据.
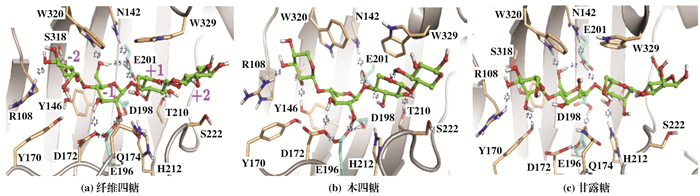
|
图 8 分子动力学模拟揭示ThCel7B的-2至+2亚位点与不同多糖的结合模式及氢键相互作用[26] Fig.8 Molecular dynamics simulation reveals the binding modes and hydrogen bonding interactions of the -2 to +2 subsites of ThCel7B with different polysaccharides[26] (a)cellotetraose; (b) xylotetraose; (c) mannotetraose |
此外, 分子动力学模拟还可用于分析决定结构稳定性与催化活性的关键因素, 用于指导糖苷酶的催化功能改造. Unal等[27]和Godoy等[28]通过分子动力学模拟计算GH45家族内切葡聚糖酶PcCel45A在3种不同温度下的RMSF值、溶剂可及表面积, 发现Asn92位点与周围氨基酸的相互作用是维持结构稳定的重要因素.在酶的分子设计中, 借助分子动力学模拟可以帮助人们解析酶活性与热稳定性的结构基础, 定位引起功能差异的关键区域, 并通过引入合适突变达到酶分子改造的目的.
4 多尺度组合模拟指导糖苷酶结构改造近年来, 量子力学(QM)及量子-分子力学组合模型(QM/MM)化学计算方法的引入, 为酶催化理论研究提供了精准的预测和验证工具.目前, 量子化学计算模型主要分为密度泛函理论(DFT)、从头算和半经验方法几类.全酶环境下的QM/MM模拟体系(图 9)通常对活性中心区域进行QM计算, 对全酶进行分子力场MM计算, 可从原子水平对糖苷酶的设计改造进行精确的计算指导, 使得人们对糖苷酶结构特征的认识达到了前所未有的原子水平[29-31].
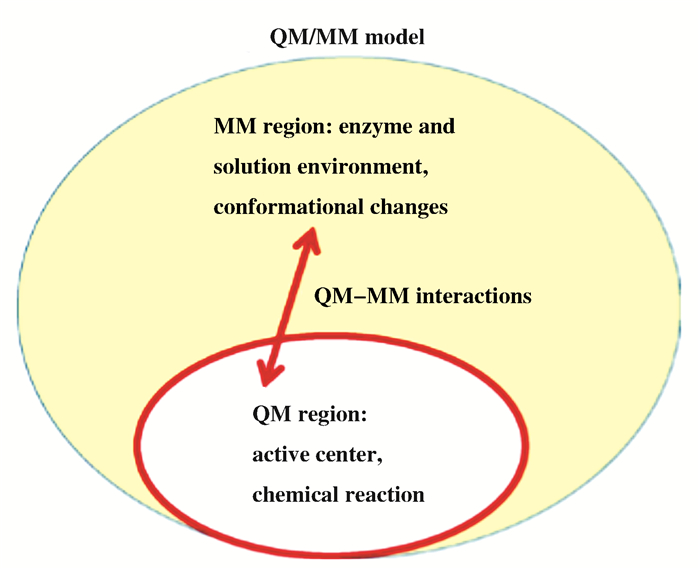
|
图 9 量子力学(QM)/分子力学(MM)模型 Fig.9 Quantum mechanics (QM)/Molecular mechanics (MM) model |
首先, QM/MM模拟在研究糖苷酶过渡态的形成机制过程中发挥了重要作用[32-35].基于DFT的QM/MM分子动力学模拟在糖苷酶催化机制研究中的应用, 加深了人们对酶催化过程中构象变化的理解, 及底物糖环的构象动力学对催化机制的影响[36].另外, 随着QM/MM多尺度模拟在糖苷酶催化机制研究中的深入, 进一步促进了糖苷酶的结构改造. Raich等[2, 37]对GH72家族β-内切葡聚糖酶与底物复合物进行QM/MM动力学模拟, 解释了底物糖基2-OH与亲核试剂(Glu275)间氢键相互作用对反
应自由能垒的影响(见图 10).通过比较具有氢键和不具有氢键的两个异构体发生反应所需的自由能, 发现当氢键相互作用去除时, 反应所需的自由能增加了11 kcal/mol.这项研究证实了2-OH与亲核试剂间的氢键对催化反应的重要作用, 为进一步设计优良酶分子提供了启示.通过QM/MM模拟跟踪酶催化过程中细微的原子运动, 可以揭示很难用实验技术捕捉到的机理细节, 指导糖苷酶进行分子设计与改造以提升其催化性能.
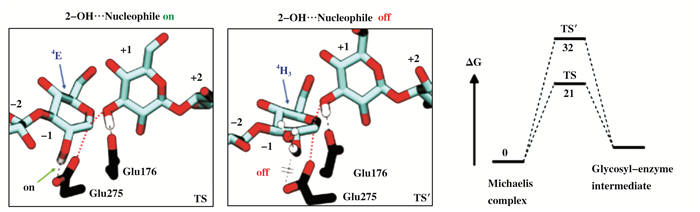
|
图 10 GH72家族β-内切葡聚糖酶的亲核试剂Glu275与2-OH氢键对过渡态自由能的影响[5, 36] Fig.10 Changes of transition-state free energy of the glycosylation reaction catalyzed by GH72 end o-β-glucanase when the 2-OH nucleophile(Glu275) interaction is switched off[5, 36] |
采用酶工程策略对天然糖苷酶进行改造是推进其工业应用的重要手段, 但如何降低酶分子改造的盲目性、提高分子设计的成功率及效率一直是亟待解决的技术难点.计算机辅助技术在糖苷酶分子设计与改造中的广泛应用, 大大提高了糖苷酶的定向改造效率, 使糖苷酶的催化性能达到了新的高度.但目前计算模拟仍存在预测精确度较差、运算速度慢、成本高的问题.随着计算机运算能力的提升和新算法在酶工程中的渗入, 计算模拟技术在糖苷酶分子设计与改造中的运用将更加便捷、成熟.这将使糖苷酶的结构预测、催化机制研究、构象动力学模拟以及分子设计改造更加理性和准确.由于不同计算分析方法具有的优势各有不同, 因此在开展酶分子设计与改造时, 结合具体改造目的而综合运用多种计算辅助方法可能会成为今后酶分子改造的重要方向.
当今, 各种计算辅助设计正在蓬勃兴起, 基于数据分析的计算设计为酶分子改造提供了新的动能[38].机器学习算法指导酶分子改造已逐渐成为酶工程新的发展趋势.相信, 随着人工智能在酶工程研究中的渗入[39], 人类将会更快捷地设计出各种满足应用需求的工业酶制剂.
| [1] |
Naumoff D G. Hierarchical classification of glycoside hydrolases[J]. Biochemistry Moscow, 2011, 76(6): 622–635.
|
| [2] |
Coines J, Raich L, Rovira C. Modeling catalytic reaction mechanisms in glycoside hydrolases[J]. Current Opinion in Chemical Biology, 2019, 53: 183–191.
|
| [3] |
Xu Y H, Feng X D, Jia J T, et al. A novel β-glucuronidase from Talaromyces pinophilus Li-93 precisely hydrolyzes glycyrrhizin into glycyrrhetinic acid 3-O-mono-β-D-glucuronide[J]. Applied and Environmental Microbiology, 2018, 84(19): e00755-18.
|
| [4] |
Li Wei-na(李伟娜), Jiang Yun-yun(蒋云云), Liu Yan-nan(刘彦楠), et al. Biocatalytic strategies in producing ginsenoside by glycosidase -a review(人参皂苷单体定向转化的生物催化及应用进展)[J]. Chinese Journal of Biotechnology(China)(生物工程学报), 2019, 35(9): 1590–1606.
|
| [5] |
Feng X D, Liu X F, Jia J T, et al. Enhancing the thermostability of β-glucuronidase from T pinophilus enables the biotransformation of glycyrrhizin at elevated temperature[J]. Chemical Engineering Science, 2019, 204: 91–98.
|
| [6] |
Singh S, Dhillon A, Goyal A. Enhanced catalytic efficiency of Bacillus amyloliquefaciens SS35 endoglucanase by ultraviolet directed evolution and mutation analysis[J]. Renewable Energy, 2020, 151: 1124–1133.
|
| [7] |
Cui H Y, Cao H, Cai H Y, et al. Computer-assisted recombination (compassr) teaches us how to recombine beneficial substitutions from directed evolution campaigns[J]. Chemistry-A European Journal, 2020, 26(3): 643–649.
|
| [8] |
Romero-Rivera A, Garcia-Borràs M, Osuna S. Computational tools for the evaluation of laboratory-engineered biocatalysts[J]. Chemical Communications, 2017, 53(2): 284–297.
|
| [9] |
Zhang Y, Aryee A N, Simpson B K. Current role of in silico approaches for food enzymes[J]. Current Opinion in Food Science, 2020, 31: 63–70.
|
| [10] |
Zheng F, Tu T, Wang X Y, et al. Enhancing the catalytic activity of a novel GH5 cellulase GtCel5 from Gloeophyllum trabeum CBS 900.73 by site-directed mutagenesis on loop 6[J]. Biotechnology for Biofuels, 2018, 11(76): 1–13.
|
| [11] |
Guerriero G, Sergeant K, Legay S, et al. Novel insights from comparative in silico analysis of green microalgal cellulases[J]. International Journal of Molecular Sciences, 2018, 19(6): 1–20.
|
| [12] |
Shen Y F, Li Z Y, Huo Y Y, et al. Structural and functional insights into CmGH1, a novel GH39 family β-Glucosidase from deep-sea bacterium[J]. Frontiers in Microbiology, 2019, 10(2922): 1–12.
|
| [13] |
Han N Y, Miao H B, Ding J M, et al. Improving the thermostability of a fungal GH11 xylanase via site-directed mutagenesis guided by sequence and structural analysis[J]. Biotechnology for Biofuels, 2017, 10(133): 1–12.
|
| [14] |
Viborg A H, Terrapon N, Lombard V, et al. A subfamily roadmap of the evolutionarily diverse glycoside hydrolase family 16(GH16)[J]. Journal of Biological Chemistry, 2019, 294(44): 15973–15986.
|
| [15] |
Pachl P, Kapešová J, Brynda J, et al. Rutinosidase from Aspergillus niger: Cystal structure and insight into the enzymatic activity[J]. The FEBS Journal, 2020, 287(15): 3315–3327.
|
| [16] |
Schiano-Di-Cola C, Kołaczkowski B, Sørensen T H, et al. Structural and biochemical characterization of a family 7 highly thermostable endoglucanase from the fungus Rasamsonia emersonii[J]. The FEBS Journal, 2020, 287(12): 2577–2596.
|
| [17] |
Lv B, Sun H L, Huang S, et al. Structure-guided engineering of the substrate specificity of a fungal β-glucuronidase toward triterpenoid saponins[J]. Journal of Biological Chemistry, 2018, 293(2): 433–443.
|
| [18] |
Li Qi(李琦), Jiang Wen-yi(蒋雯怡), Sun Yu-wei(孙雨薇), et al. Thermostability modification of the GH11 xylanase MxynB from Aspergillus niger NL-1 and its application in xylooligosaccharide production(Aspergillus niger NL-1来源的木聚糖酶的耐热性能改造及其在水解木聚糖中的应用)[J]. Chemical Industry and Engineering Progress(China)(化工进展), 2019, 38(2): 1038–1044.
|
| [19] |
Cui Ying-lu(崔颖璐), Wu Bian(吴边). A brief overview of computational protein structure prediction and enzyme design(计算机辅助蛋白结构预测及酶的计算设计研究进展)[J]. Guangxi Sciences(China)(广西科学), 2017, 24(1): 1–6.
|
| [20] |
Dadheech T, Jakhesara S, Chauhan P S, et al. Draft genome analysis of lignocellulolytic enzymes producing Aspergillus terreus with structural insight of β-glucosidases through molecular docking approach[J]. International Journal of Biological Macromolecules, 2019, 125: 181–190.
|
| [21] |
Zhang X L, Sitasuwan P, Horvath G, et al. Increased activity of β-glucuronidase variants produced by site-directed mutagenesis[J]. Enzyme and Microbial Technology, 2018, 109: 20–24.
|
| [22] |
Shanmugam R, Kim I W, Tiwari M K, et al. Tyr320 is a molecular determinant of the catalytic activity of β-glucosidase from Neosartorya fischeri[J]. International Journal of Biological Macromolecules, 2020, 151: 609–617.
|
| [23] |
Ebert M C C J C, Espinola J G, Lamoureux G, et al. Substrate-specific screening for mutational hotspots using biased molecular dynamics simulations[J]. ACS Catalysis, 2017, 7(10): 6786–6797.
|
| [24] |
Chan H C S, Pan L, Li Y, et al. Rationalization of stereoselectivity in enzyme reactions[J]. Wiley Interdisciplinary Reviews: Computational Molecular Science, 2019, 9(4): 1–17.
|
| [25] |
Liu B B, Qu G, Li J K, et al. Conformational dynamics-guided loop engineering of an alcohol dehydrogenase: Capture, turnover and enantioselective transformation of difficult-to-reduce ketones[J]. Advanced Synthesis & Catalysis, 2019, 361(13): 3182–3190.
|
| [26] |
Sonoda M T, Godoy A S, Pellegrini V O A, et al. Structure and dynamics of Trichoderma harzianum Cel7B suggest molecular architecture adaptations required for a wide spectrum of activities on plant cell wall polysaccharides[J]. Biochimica et Biophysica Acta (BBA) - General Subjects, 2019, 1863(6): 1015–1026.
|
| [27] |
Unal M A, Boyacioglu B, Unver H, et al. Molecular simulation of PcCel45A protein expressed from Aspergillus nidulans to understand its structure, dynamics, and thermostability[J]. Journal of Molecular Modeling, 2019, 25(11): 1–7.
|
| [28] |
Godoy A S, Pereira C S, Ramia M P, et al. Structure, computational and biochemical analysis of PcCel45A endoglucanase from Phanerochaete chrysosporium and catalytic mechanisms of GH45 subfamily C members[J]. Scientific Reports, 2018, 8(3678): 1–14.
|
| [29] |
Liu Hai-yan(刘海燕). Computational chemistry approaches in studies on industrial enzymes(工业酶研究中的计算化学方法)[J]. Chinese Journal of Biotechnology(China)(生物工程学报), 2019, 35(10): 1819–1828.
|
| [30] |
Wang B J, Walton P H, Rovira C. Molecular mechanisms of oxygen activation and hydrogen peroxide formation in lytic polysaccharide monooxygenases[J]. ACS Catalysis, 2019, 9(6): 4958–4969.
|
| [31] |
Chen N H, Wang S L, Smentek L, et al. Biosynthetic mechanism of lanosterol: Cyclization[J]. Angewandte Chemie International Edition, 2015, 54(30): 8693–8696.
|
| [32] |
Bottoni A, Miscione G P, Vivo M D. A theoretical DFT investigation of the lysozyme mechanism: Computational evidence for a covalent intermediate pathway[J]. Proteins: Structure, Function, and Bioinformatics, 2005, 59(1): 118–130.
|
| [33] |
Withers S G, Warren R A J, Street I P, et al. Unequivocal demonstration of the involvement of a glutamate residue as a nucleophile in the mechanism of a retaining glycosidase[J]. Journal of the American Chemical Society, 1990, 112(15): 5887–5889.
|
| [34] |
White A, Tull D, Johns K, et al. Crystallographic observation of a covalent catalytic intermediate in a beta-glycosidase[J]. Nature Structural Biology, 1996, 3(2): 149–154.
|
| [35] |
Bowman A L, Grant I M, Mulholland A J. QM/MM simulations predict a covalent intermediate in the hen egg white lysozyme reaction with its natural substrate[J]. Chemical Communications (Cambridge, England), 2008, 2008(37): 4425–4427.
|
| [36] |
Ardèvol A, Rovira C. Reaction mechanisms in carbohydrate-active enzymes: Glycoside hydrolases and glycosyltransferases. Insights from ab initio quantum mechanics/molecular mechanics dynamic simulations[J]. Journal of the American Chemical Society, 2015, 137(24): 7528–7547.
|
| [37] |
Raich L, Borodkin V, Fang W, et al. A trapped covalent intermediate of a glycoside hydrolase on the pathway to transglycosylation. Insights from experiments and quantum mechanics/molecular mechanics simulations[J]. Journal of the American Chemical Society, 2016, 138(10): 3325–3332.
|
| [38] |
Mazurenko S, Prokop Z, Damborsky J. Machine learning in enzyme engineering[J]. ACS Catalysis, 2020, 10(2): 1210–1223.
|
| [39] |
Chen K, Arnold F H. Engineering new catalytic activities in enzymes[J]. Nature Catalysis, 2020, 3(3): 203–213.
|
 2020, Vol. 34
2020, Vol. 34


