手性是自然界的本质属性之一, 作为生命活动重要物质基础的生物大分子几乎全是手性的. 手性化合物由于其独特的理化特性与生物活性, 近年来在新材料开发、新药设计和精细化学品合成等领域具有越来越广泛的应用[1]. 手性化合物的传统制备方法是化学催化法, 采用化学法易造成环境污染、反应条件苛刻等缺点, 难以满足实际工业生产的需求[2]. 与传统的化学法相比, 生物催化法因具有选择性强、反应条件温和、环境友好等优点而在生产中深受青睐[3].
目前, 手性化合物外消旋体的生物催化去对称化已成为获得单一手性对映体的主要途径之一[4]. 我们结合近年来文献报道情况, 将近年来发展起来的手性化合物生物催化去对称化方法归纳为立体转化去对称化、线性去对称化、循环去对称化、对映体收敛去对称化、一步去对称化5大类, 下面对这些生物催化去对称化方法的特点及其具体应用进展情况分别进行介绍.
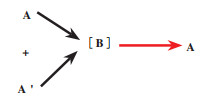
1 立体转化去对称化生物催化立体转化去对称化是利用生物催化剂—酶或细胞催化的反应, 将外消旋底物中的一种对映体A’转化为前手性中间体B, 该中间体再被进一步转化为另一种对映体A[5](图 1). 其原理在于通过破坏一种对映体的原有手性中心后重新构建新的手性中心, 将其转化为另一种对映体, 从而达到去对称化的目的.
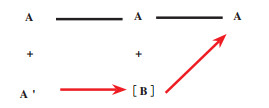
|
图 1 立体转化去对称化 Fig.1 Desymmetrization by stereoinversion (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
酶催化立体转化去对称化是利用两种或多种不同酶催化外消旋底物中的一种对映体, 依次进行多步转化反应后, 生成另一种对映体, 例如外消旋醇、氨基酸以及胺类化合物的立体转化去对称化.
外消旋醇、氨基酸可通过先形成羰基中间体再经不对称还原而实现去对称化. 例如Nie等[6]依次通过羰基还原酶1(SCR1)选择性氧化、酮还原酶(KRED)选择性还原, 将外消旋1-苯基-1, 2-乙二醇转化为(R)-1-苯基-1, 2-乙二醇(图 2), ee值为95.5%, 转化率为91.6%. Han等[7]依次通过氨基酸脱氢酶催化的选择性氧化反应和ω-转氨酶催化的不对称还原反应, 将D, L-丙氨酸转化为D-丙氨酸(图 3), 转化率为95.0%, ee值> 99.0%.
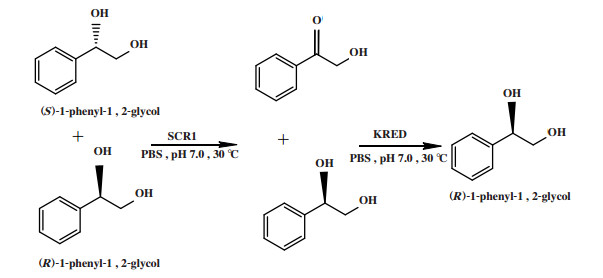
|
图 2 外消旋1-苯基-1, 2-乙二醇的立体转化去对称化 Fig.2 Desymmetrization by stereoinversion of racemic 1-phenyl-1, 2-glycol |
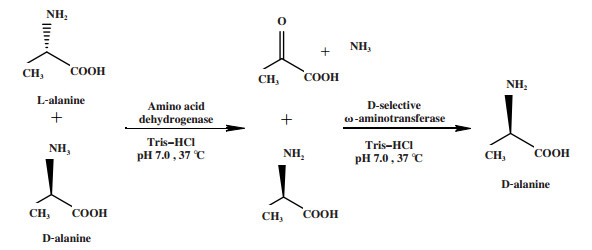
|
图 3 DL-丙氨酸的立体转化去对称化 Fig.3 Desymmetrization by stereoinversion of racemic alanine |
外消旋胺类化合物则可通过先形成亚胺基中间体再经不对称还原而实现去对称化. 例如Li等[8]利用D-氨基酸氧化酶(FsDAAO)氧化(R)-1-羧基-1, 2, 3, 4-四氢异喹啉得到亚胺基中间体, 再利用羧酸还原酶(PpDpkA)对亚胺基中间体进行还原, 得到具有高转化率(> 99.0%)和对映体过量(> 99.0%)的(S)-1-羧基-1, 2, 3, 4-四氢异喹啉(图 4).
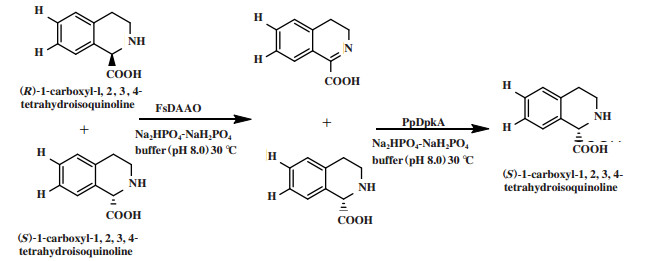
|
图 4 外消旋1-羧基-1, 2, 3, 4-四氢异喹啉的立体转化去对称化 Fig.4 Desymmetrization by stereoinversion of racemic 1-carboxyl-1, 2, 3, 4-tetrahydroisoquinoline |
酶催化立体转化去对称化的多步反应可在同一个反应器内进行, 即通过一锅级联反应将外消旋体转化为单一手性对映体[9], 从而省去了中间产物分离的步骤. 同时, 可通过反应器内选择性氧化酶与选择性还原酶的自偶联作用而实现辅因子的再生, 从而大幅降低成本.
1.2 化学/酶催化立体转化去对称化化学/酶催化立体转化去对称化是将化学催化与酶催化联用协同催化外消旋底物去对称化. 例如Rosini等[10]利用重组变形杆菌L-氨基酸脱氨酶(PmaLAAD)选择性催化L-苯丙氨酸脱氢后, 再以硼烷三乙胺配合物作为化学还原剂, 选择性还原生成D-苯丙氨酸(图 5), 转化率为70.0%, ee值> 99.0%.
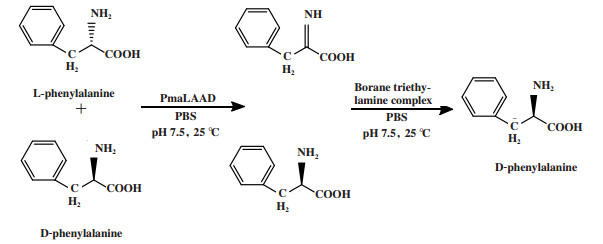
|
图 5 DL-苯丙氨酸立体转化去对称化 Fig.5 Desymmetrization by stereoinversion of racemic phenylalanine |
全细胞催化立体转化去对称化是通过微生物全细胞或微生物全细胞与酶联用, 协同催化外消旋底物立体转化去对称化. 例如Nasario等[11]采用白色念珠菌Candida albicans CCT 0776和酮还原酶联用的方法, 催化外消旋1-苯乙醇进行立体转化去对称化, 得到(R)-1-苯乙醇(图 6), 转化率为90.0%, ee值为83.0%.
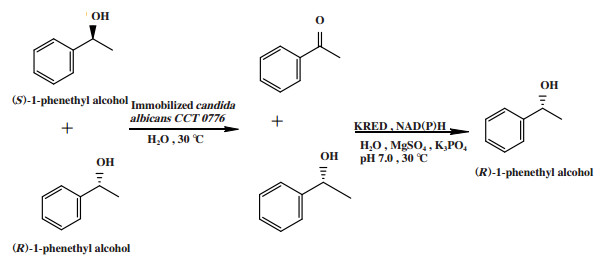
|
图 6 外消旋1-苯乙醇的立体转化去对称化 Fig.6 Desymmetrization by stereoinversion of racemic 1-phenethyl alcohol |
通过选择性氧化/还原级联反应催化外消旋底物去对称化还可以在大肠杆菌等工程细胞中多基因共表达来实现[12]. 例如Zheng等[13]通过重组大肠杆菌工程菌株共表达(S)-2-羟酸脱氢酶(HADH)、(R)-2-酮酸还原酶(LlKAR)和葡萄糖脱氢酶(GDH), 构建了外消旋2-羟基苯乙酸氧化/还原偶联去对称化及辅酶再生系统. 该系统利用(S)-2-羟酸脱氢酶将(S)-2-羟基苯乙酸选择性氧化为酮酸, 所得的酮酸随后被(R)-2-酮酸还原酶选择性还原为(R)-2-羟基苯乙酸(图 7), 得到(R)-2-羟基苯乙酸, 转化率为98.5%, ee值> 99.0%.
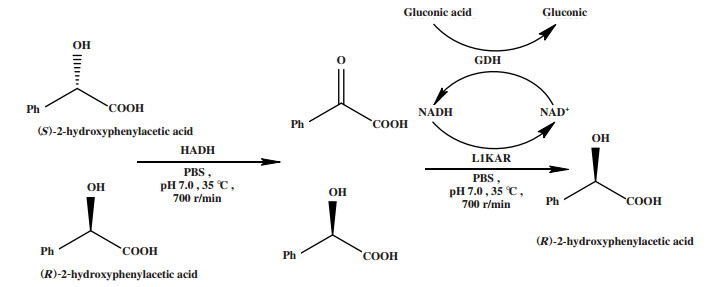
|
图 7 外消旋2-羟基苯乙酸的立体转化去对称化 Fig.7 Desymmetrization by stereoinversion of racemic 2-hydroxyphenylacetic acid |
这种新型全细胞生物催化剂通过对多种酶的共表达, 实现胞内的辅酶再生, 无需添加辅酶, 反应更环保、成本更低, 因此在实际应用中更具前景和竞争力.
2 线性去对称化线性去对称化通常是先将外消旋底物A和A’非选择性转化为前手性中间体B, 然后该前手性中间体再被选择性转化为单一手性对映体A[14](图 8). 线性去对称化与立体转化去对称化的不同之处在于前者是外消旋底物的两种对映体均被转化为同一中间体. 其原理在于通过破坏两种对映体的原有手性中心后, 重新构建新的手性中心, 将其转化为单一手性对映体, 从而达到去对称化的目的.
|
图 8 线性去对称化 Fig.8 Linear desymmetrization (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
酶催化线性去对称化全过程均使用酶作为催化剂催化外消旋底物去对称化. 例如Masateru等[15]使用来自嗜热厌氧乙醇菌的醇脱氢酶的单一突变体(W110G TESADH)催化外消旋4-对羟基苯基-2-丁醇非选择性氧化得到中间体酮, 然后利用该酶催化中间体酮进行选择性还原, 得到(S)-4-对羟基苯基-2-丁醇(图 9), 转化率 > 99.5%, ee值为99.0%.
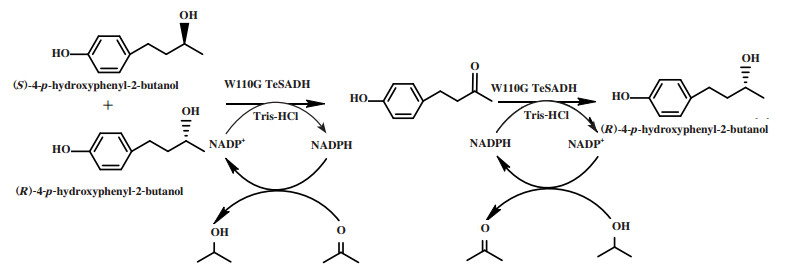
|
图 9 外消旋4-对羟基苯基-2-丁醇的线性去对称化 Fig.9 Linear desymmetrization of racemic 4-p-hydroxyphenyl-2-butanol |
李伟翔[16]使用乙醇脱氢酶(ADH-A3263)催化外消旋2-庚醇非选择性氧化得到中间体酮, 再利用酮还原酶(E-Ketoreductase)选择性还原中间体酮得到(R)-2-庚醇(图 10), 成功实现了外消旋2-庚醇的去对称化. 赵依威[17]通过氧化还原双酶偶联催化外消旋1-苯乙醇的去对称化, 得到(R)-1-苯乙醇(图 11).
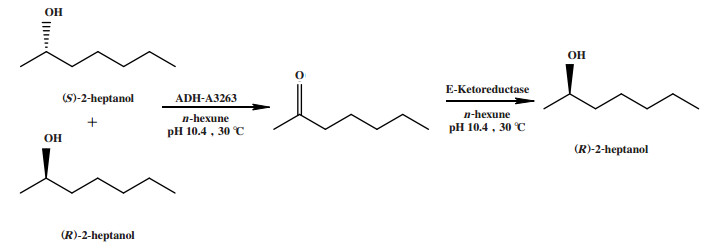
|
图 10 外消旋2-庚醇的线性去对称化 Fig.10 Linear desymmetrization of racemic 2-heptanol |
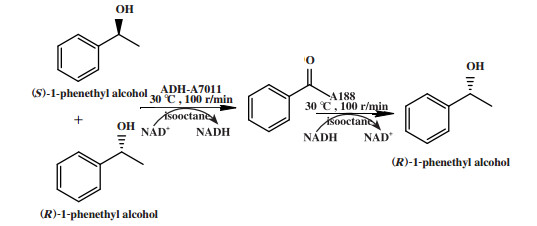
|
图 11 外消旋1-苯乙醇的线性去对称化 Fig.11 Linear desymmetrization of racemic 1-phenethyl alcohol |
化学/酶催化线性去对称化是将酶与化学催化剂联用, 协同催化外消旋底物去对称化而得到单一手性对映体. 例如Moris等[18]利用有机催化剂2-氮杂金刚烷N-氧基(AZADO)将外消旋1-苯甲基乙醇非选择性氧化得到中间体酮, 并在同一个反应器内利用酮还原酶(P3-B03)将其选择性还原为(S)-1-苯甲基乙醇(图 12), 转化率为98.0%, ee值> 99.0%.
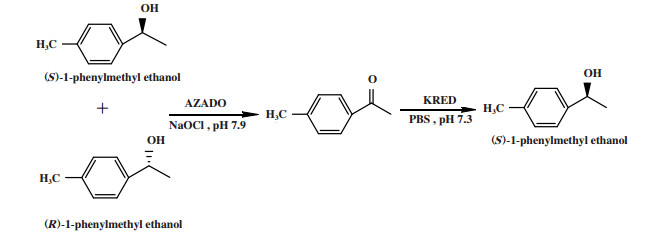
|
图 12 外消旋1-苯甲基乙醇线性去对称化 Fig.12 Linear desymmetrization of racemic 1-phenylmethyl ethanol |
化学/酶催化线性去对称化可在一个反应器内进行, 但需考虑化学催化剂与酶的接触失活问题, 可通过介孔有机二氧化硅、MOF材料、聚二甲基硅氧烷膜等材料作为载体进行酶的固定化, 使酶与化学催化剂分隔开[19-20], 以避免化学催化剂与酶之间的相互干扰.
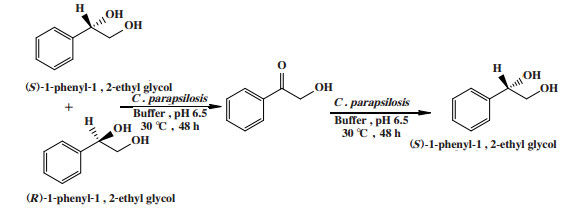
2.3 全细胞催化线性去对称化全细胞催化线性去对称化是指利用全细胞作为催化剂, 催化外消旋底物进行线性去对称化而得到单一手性对映体. 例如Chadha等[21]通过假丝酵母菌共表达醇脱氢酶、羰基还原酶实现外消旋1-苯基-1, 2-乙二醇的线性去对称化. 外消旋1-苯基-1, 2-乙二醇利用醇脱氢酶非选择性氧化为中间体酮, 该中间体再被羰基还原酶选择性还原为(S)-1-苯基-1, 2-乙二醇(图 13), 转化率高达92.0%, ee值为98.0%.
|
图 13 外消旋1-苯基-1, 2-乙二醇线性去对称化 Fig.13 Linear desymmetrization of racemic 1-phenyl-1, 2-ethylene glycol |
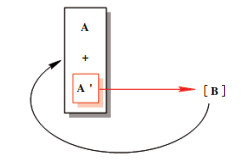
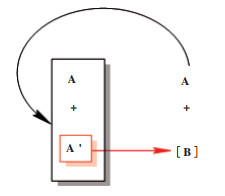
循环去对称化有两种形式. 循环去对称化1是将外消旋底物A和A’中的一个对映体A’选择性转化为中间体B, 再将B非选择性转化为外消旋底物A和A’, 然后将对映体A’选择性转化为B, 从而构成一个循环. 经过多轮循环后, 可得到单一手性对映体A[22](图 14). 通过每次循环过程中一种对映体的消耗和另一种对映体的积累, 从而达到去对称化的目的. 循环去对称化2是将外消旋底物A和A’中的一个对映体A’选择性转化为中间体B, 再将另一个对映体A非选择性转化为外消旋底物A和A’, 然后将对映体A’选择性转化为B, 从而构成一个循环, 经过多轮循环后, 可得到单一手性对映体B[23](图 15).
|
图 14 循环去对称化1 Fig.14 Cyclic desymmetrization 1 (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
|
图 15 循环去对称化2 Fig.15 Cyclic desymmetrization 2 (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
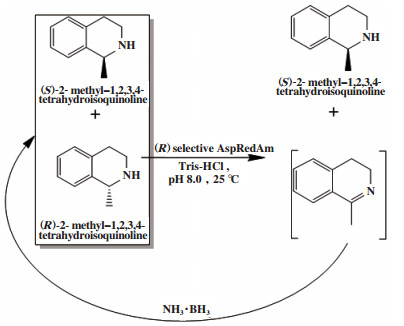
外消旋胺类化合物的循环去对称化1通常是通过胺氧化酶催化外消旋底物选择性氧化和氨硼烷或硼氢化钠等化学催化剂对亚胺中间体进行非选择性还原实现的. 例如Heath等[24]开发了一个可再生辅因子的循环去对称化系统, 利用米曲霉中NADP(H)依赖的还原性氨化酶(AspRedAm)氧化(R)-2-甲基-1, 2, 3, 4-四氢异喹啉得到亚胺中间体, 该中间体再被氨硼烷非选择性催化(图 16), 得到(S)-2-甲基-1, 2, 3, 4-四氢异喹啉, 转化率达到98.0%, ee值> 99.0%.
|
图 16 外消旋2-甲基-1, 2, 3, 4-四氢异喹啉循环去对称化 Fig.16 Cyclic desymmetrization of racemic 2-methyl-1, 2, 3, 4-tetrahydroisoquinoline |
光学纯手性杂环化合物-哌啶类、吗啡啉类和哌嗪类是抗组胺类药物的重要前体, 例如咪唑斯汀、西替利嗪、布可利嗪等. Pontini等[25]利用来自黑曲霉的单胺氧化酶(MAO-N)选择性氧化(S)-2-苯基哌啶, 得到亚胺中间体, 该中间体再被氨硼烷非选择性催化(图 17), 得到(R)-2-苯基哌啶.
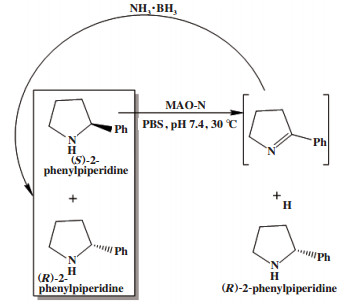
|
图 17 外消旋2-苯基哌啶循环去对称化 Fig.17 Cyclic desymmetrization of racemic 2-phenylpiperidine |
光催化条件温和, 与生物催化相结合可实现外消旋底物的高效循环去对称化. 例如Schreier等[26]利用MAO-N-9选择性氧化(S)-2-正丁基环戊胺得到亚胺中间体, 该中间体再被[Ru(bpy)3]Cl2作为光催化剂、可见光驱动(AscH2)作为还原剂进行非选择性催化(图 18), 得到(R)-2-正丁基环戊胺, 转化率 > 95.0%, ee值为98.0%.
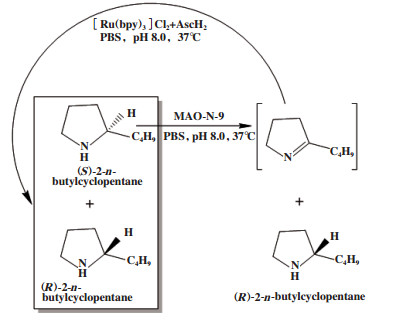
|
图 18 外消旋2-正丁基环戊烷循环去对称化 Fig.18 Cyclic desymmetrization of racemic 2-n-butylcyclopentane |
外消旋胺类化合物的循环去对称化2通常是通过脂肪酶对外消旋底物选择性催化和化学催化剂消旋实现的. 例如Ferraz等[27]利用来源于南极假丝酵母的脂肪酶B(CALB)选择性催化(R)-1-苯乙胺得到(R)-2-甲氧基-N-(1-苯乙基)乙酰胺, (S)-1-苯乙胺被钯催化剂消旋(图 19), 得到(S)-2-甲氧基-N-(1-苯乙基)乙酰胺, 转化率达到95.0%, ee值> 99.0%.
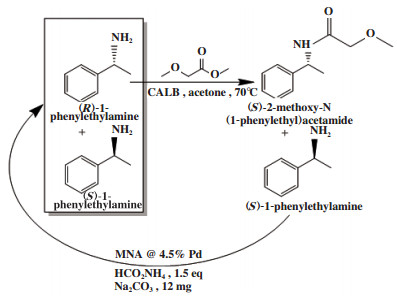
|
图 19 外消旋1-苯乙胺循环去对称化 Fig.19 Cyclic desymmetrization of racemic 1-phenylethylamine |
外消旋醇类化合物的循环去对称化1通常是通过酶/微生物细胞对外消旋底物选择性氧化和化学催化剂对酮中间体非选择性还原, 经多轮循环得到单一手性醇. 例如Magallanes-Noguera等[28]通过固定化贾斯米德菌G. jasminoides选择性氧化、硼氢化钠非选择性还原, 催化外消旋1-苯乙醇循环去对称化, 得到(R)-1-苯乙醇, 转化率为84.0%, ee值为97.0%(图 20).
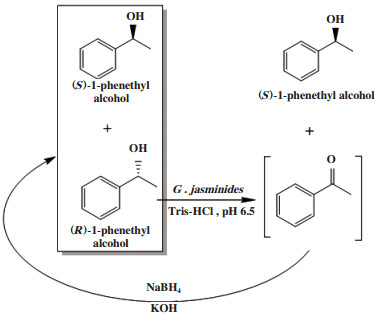
|
图 20 外消旋1-苯乙醇循环去对称化 Fig.20 Cyclic desymmetrization of racemic 1-phenethyl alcohol |
循环去对称化1还可通过选择性还原和非选择性氧化结合, 以此得到目标手性对映体. 例如Nosek等[29]将外消旋亚砜中的(S)-对映体经甲硫氨酸硫氧化物还原酶(MsrA)选择性还原得到非手性硫化物中间体, 再经恶唑烷氧化剂非选择性氧化得到外消旋亚砜. 循环反应24 h, 得到(R)-亚砜(图 21), 转化率为90.0%, ee值> 99.0%.
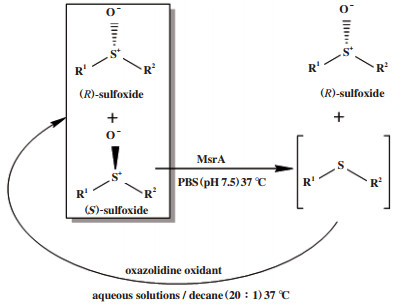
|
图 21 外消旋亚砜循环去对称化 Fig.21 Cyclic desymmetrization of racemic sulfoxide |
外消旋醇类化合物的循环去对称化2通常是通过脂肪酶对外消旋底物选择性氧化、化学催化剂消旋, 经多轮循环得到单一手性酯. 例如Yun等[30]利用CALB选择性催化(S)-1-苯乙醇得到(S)-1-乙酸苯乙酯, (R)-1-苯乙醇被钌催化剂消旋(图 22), 得到(S)-1-乙酸苯乙酯, 转化率达到96.0%, ee值为99.0%.
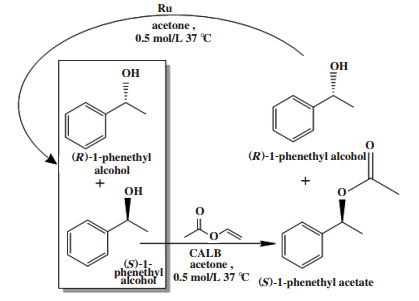
|
图 22 外消旋1-苯乙醇循环去对称化 Fig.22 Cyclic desymmetrization of racemic 1-phenethyl alcohol |
对映体会聚去对称化是将外消旋底物A和A’经过不同的生物催化过程, 最终得到同一种构型的单一手性对映体E[31](图 23). 该方法可分为一步会聚、多步会聚和混合会聚.
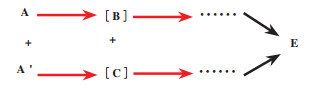
|
图 23 对映体会聚去对称化 Fig.23 Enantioconvergent process desymmetrization (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
一步对映体会聚去对称化是通过酶或全细胞催化外消旋底物转化时, 两种对映体中的一种对映体以原构型保留, 而另一种对映体构型发生翻转, 从而得到同一构型对映体. 例如Schober等[32]将外消旋(S)-磺酸酯与铜绿假单胞菌硫酸酯酶(PAS)反应, (R)-磺酸酯与波罗的海假单胞菌硫酸酯酶(PISA1)反应, 最终得到单一手性对映体仲醇(图 24), 转化率为82.0%~90.0%, ee值为95.0%~99.0%.
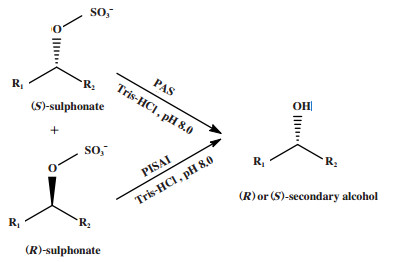
|
图 24 外消旋磺酸酯的对映体会聚去对称化 Fig.24 Enantioconvergent process desymmetrization of racemic sulphonate |
一步对映体会聚去对称化也可通过化学/酶催化剂分别选择性催化外消旋底物的两种不同对映体来实现, 例如Wu等[33]将外消旋对硝基苯乙烯氧化物的(S)-对映体与环氧水解酶2(VrEH2)反应, (R)-对映体与环氧水解酶1(VrEH1)反应, 得到(R)-对硝基苯基乙二醇(图 25), 转化率为71.5%, ee值为99.0%.
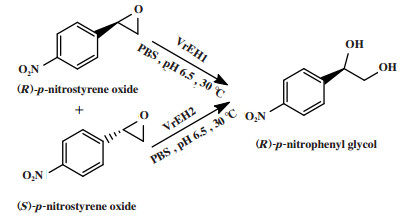
|
图 25 外消旋对硝基苯乙烯氧化物的对映体会聚去对称化 Fig.25 Enantioconvergent process desymmetrization of racemic p-nitrostyrene oxide |
多步对映体会聚去对称化是外消旋底物的两种不同对映体分别经过两个或多个反应途径转化, 最终得到相同构型的单一手性对映体. 例如Palombo等[34]对外消旋3-甲基-3, 3-二羟基环己酮的(R)-对映体经酯化、消去、水解, (S)-对映体经甲磺酰化、酯化、水解, 均可得到(R)-4-羟基-3-甲基-2-环烯酮(图 26), 转化率为65.0%, ee值为95.0%.
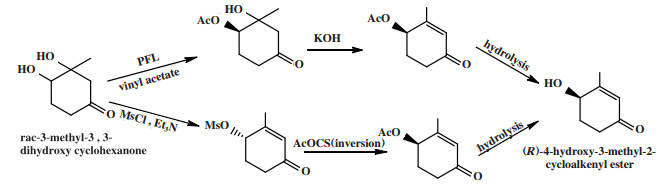
|
图 26 外消旋3-甲基-3, 3-二羟基环己酮的对映体会聚去对称化 Fig.26 Enantioconvergent process desymmetrization of racemic 3-methyl-3, 3-dihydroxy cyclohexanone |
一步去对称化是指通过酶、全细胞或化学催化剂催化, 一步实现外消旋底物的去对称化(图 27), 以消旋酶催化的去对称化最为常见. 消旋酶催化去对称化是外消旋底物A和A’经消旋酶催化得到单一手性对映体A或A’[35], 反应过程相对简单.

|
图 27 一步去对称化 Fig.27 One-step desymmetrization (红色箭头代表该步骤为选择性反应) (The red arrow indicates that this step is a selective reaction) |
例如Xu等[36]利用扁桃酸消旋酶(MR)催化外消旋扁桃酸, 一步去对称化得到D-扁桃酸(图 28). D-扁桃酸经D-扁桃酸脱氢酶(DMDH)、L-亮氨酸脱氢酶(L-LeuDH)得到L-苯基甘氨酸.
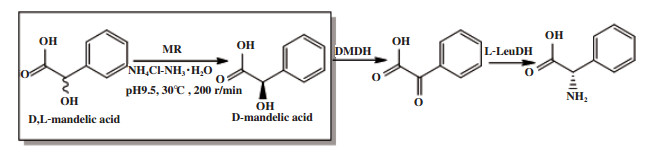
|
图 28 DL-扁桃酸的一步去对称化 Fig.28 One-step desymmetrization of racemic mandelic acid |
消旋酶催化去对称化不需要辅因子, 仅需一步反应就可得到单一对映体, 是所有去对称化方法中最简单有效的方法. 但是天然消旋酶的种类非常有限, 很难从自然界中筛选得到, 从而限制了该方法在生产中的应用.
6 展望生物催化因其绿色环保、条件温和等优点在手性合成中而深受青睐. 手性化合物生物催化去对称化方法众多, 在具体应用中各有利弊. 例如化学/酶法催化外消旋底物立体转化去对称化可将酶催化与化学催化二者的优势充分结合, 实现单一手性化合物的高效制备[37]. 但是同釜反应时通常会导致催化剂接触失活. 利用蛋白支架将有机金属催化剂包埋形成人工金属酶是解决催化剂接触失活的有效方法之一[38], 也可利用膜分隔技术将两种催化剂分别限制在膜的两侧, 以达到催化剂空间上的分隔[39]. 在线性去对称化中, 全细胞催化外消旋底物线性去对称化可以实现辅因子自再生, 但是会因细胞壁和细胞膜的传质障碍而影响酶的催化活性, 可以利用细胞透性化技术对细胞壁和细胞膜的结构进行改造以降低其对传质与扩散的不良影响[40]. 在前述5大生物催化去对称化方法中, 以消旋酶催化外消旋底物的直接去对称化最为简单有效, 但是目前已知的消旋酶种类非常有限, 且难以从自然界中直接筛选得到作用于特定手性底物的消旋酶. 未来有望通过对已知消旋酶作用机制的深入研究, 利用酶的人工模拟与理性设计等方法来获得满足生产需要的人工消旋酶, 实现外消旋底物高效去对称化[41].
生物催化酶促去对称化方法中存在的共性问题是如何提高酶的立体选择性, 可以通过定向进化[42]、理性设计[43]、DNA改组[44]等方法改造酶分子构象, 使酶的选择性大幅提高. 另外, 涉及到氧化还原反应的去对称化方法中, 辅因子再生是一个亟待解决的技术难点, 可通过酶法、化学法、电化学法和光化学法等实现辅因子再生[45-46], 目前, 研究和应用最多的是酶法再生, 其中甲酸/甲酸脱氢酶(FDH)体系是较成功的再生系统, 并已实现工业生产应用. 其优势在于反应不可逆, 只产生CO2一种副产物[47].
在可预见的未来, 随着合成生物技术、蛋白质与酶工程以及化学与化工等不同领域的发展与交叉融合, 手性化合物生物催化去对称化技术将会得到进一步发展和完善, 生物催化去对称化的方法会更丰富、更高效, 在工业生产中的应用也会越来越广泛.
| [1] |
a. Li Wei-xiang(李伟翔), Li Chun(李春), Liu Gui-yan(刘桂艳), et al. Advances in enzymatic asymmetric catalytic chiral synthesis systems(酶促不对称催化手性合成系统研究进展)[J]. J Mol Catal(China)(分子催化), 2014, 28(6): 581-594. b. Wei Zhao(魏钊), Yang Hua-meng(杨化萌), Zhang Jin-long(张金龙), et al. Direct onstruction of axially chiral 4-arylquinoline via acid-promoted dehydrative cyclization/oxidation of diarylmethanols(酸催化的二芳基甲醇脱水环化氧化芳构化直接构筑4-芳基喹啉)[J]. J Mol Catal(China)(分子催化), 2019, 33(4): 371-381. c. Zhang An-lin(张安林), Xu Hu(许慧), Pan Chun-yue(潘春跃), et al. Highly nantioselective asymmetric epoxidation of olefins catalyzed by chiral polyethers(手性聚醚催化烯烃的高对映选择性环氧化反应研究)[J]. J Mol Catal(China)(分子催化), 2020, 34(6): 514-524. |
| [2] |
Zheng G W, Xu J H, Turner N J, et al. Enantioselective synthesis of chiral vicinal amino alcohols using amine dehydrogenases[J]. ACS Catal, 2019, 9(12): 11813–11818.
DOI:10.1021/acscatal.9b03889 |
| [3] |
Xia Shi-wen(夏仕文), Xiong Wen-juan(熊文娟), Wei Yan-chan(韦燕婵), et al. Advances in the amino acid oxidase-catalyzed synthesis of non-natural chiral amino acids(氨基酸氧化酶催化合成非天然手性氨基酸研究进展)[J]. J Mol Catal (China)(分子催化), 2015, 29(3): 288–298.
|
| [4] |
Azofra L M, Tran M A, Zubar V, et al. Conversion of racemic alcohols to optically pure amine precursors enabled by catalyst dynamic kinetic resolution: Experiment and computation[J]. Chem Commun, 2020, 56(64): 9094–9097.
DOI:10.1039/D0CC02881A |
| [5] |
Palomo J M. Semisynthetic enzymes by protein-peptide site-directed covalent conjugation: Methods and applications[J]. Method Enzymol, 2017, 590: 305–316.
|
| [6] |
Nie Y, Mu X Q, Xu Y, et al. De novo construction of multienzyme system for one-pot deracemization of (R, S)-1-phenyl-1, 2-ethanediol by stereoinversion of (S)-enantiomer to the corresponding counterpart[J]. J Mol Catal B-Enzym, 2016, 129: 21–28.
DOI:10.1016/j.molcatb.2016.04.003 |
| [7] |
Han S W, Shin J S. One-pot preparation of d-amino acids through biocatalytic deracemization using alanine dehydrogenase and ω-transaminase[J]. Catal Lett, 2018, 148(12): 3678–3684.
DOI:10.1007/s10562-018-2565-3 |
| [8] |
Li J, Yang L R, Wu J P, et al. A biocatalytic redox cascade approach for one-pot deracemization of carboxyl-substituted tetrahydroisoquinolines by stereoinversion[J]. Green Chem, 2019, 21(20): 5579–5585.
DOI:10.1039/C9GC02795E |
| [9] |
Ying Xiang-xian(应向贤), Mao Wang-wei(毛王伟), Zhang Li(张丽), et al. Multi-enzymatic cascade ractions involving oxidoreductases(氧化还原酶在多酶级联反应中的应用进展)[J]. Bull Fermen Sci Technol(China)(发酵科技通讯), 2018, 47(3): 129–133.
|
| [10] |
Rosini E, Melis R, Molla G, et al. Deracemization and stereoinversion of α-amino acids by L-amino acid deaminase[J]. Adv Synth Catal, 2017, 359(21): 3373–3381.
|
| [11] |
Nasario F D, Moran P J S, Rodrigues J A R. Deracemization of sec-alcohols through sequential application of C. Albicans and ketoreductases[J]. J Brazil Chem Soc, 2019, 30(4): 772–779.
|
| [12] |
Bayer T, Milker S, Wiesinger T, et al. Designer microorganisms for optimized redox cascade reactions-challenges and future perspectives[J]. Adv Synth Catal, 2015, 357(8): 1587–1618.
DOI:10.1002/adsc.201500202 |
| [13] |
Zheng Y G, Wang D C, Liu Z Q, et al. Highly efficient deracemization of racemic 2-hydroxy acids in a three-enzyme co-expression system using a novel ketoacid reductase[J]. Appl Biochem Biotechol, 2018, 186(3): 563–575.
DOI:10.1007/s12010-018-2760-0 |
| [14] |
Moris F, Gonzalez-Sabin J, Berglund P, et al. Chemoenzymatic synthesis of sertraline[J]. Eur J Org Chem, 2020, 2020(4): 510–513.
DOI:10.1002/ejoc.201901810 |
| [15] |
Masateru T, Hamdan S M, Musa M M, et al. Deracemization of secondary alcohols by using a single alcohol dehydrogenase[J]. ChemCatChem, 2016, 8(8): 1459–1463.
DOI:10.1002/cctc.201600160 |
| [16] |
Li Wei-xiang(李伟翔). Directed control of enzymatic stereoselectivity in biocatalytic asymmetric catalysis(生物不对称催化中酶立体选择性的定向调控)[D]. Beijing(北京): Beijing Instisute of Technology(北京理工大学), 2015.
|
| [17] |
Zhao Yi-wei(赵依威). Asymmetric synthesis of (R)-1phenylethanol catalyzed by the coupling of oxidoreductases(氧化还原双酶偶联催化不对称合成(R)-1-苯乙醇))[D]. Beijing(北京): Beijing Instisute of Technology(北京理工大学), 2021.
|
| [18] |
Moris F, Gonzalez-Sabin J, Rebolledo F, et al. A straightforward deracemization of sec-alcohols combining organocatalytic oxidation and biocatalytic reduction[J]. Eur J Org Chem, 2018, 2018(23): 3031–3035.
DOI:10.1002/ejoc.201800569 |
| [19] |
Mohammadi N S, Khiabani M S, Ghanbarzadeh B, et al. Improvement of lipase biochemical properties via a two-step immobilization method: Adsorption onto silicon dioxide nanoparticles and entrapment in a polyvinyl alcohol/alginate hydrogel[J]. J Biotechnol, 2020, 323: 189–202.
DOI:10.1016/j.jbiotec.2020.07.002 |
| [20] |
Zou Y L, Liu X Y, Zhang H X. A dual enzyme-containing microreactor for consecutive digestion based on hydrophilic ZIF-90 with size-selective sheltering[J]. Colloid Surface B, 2021, 197: 111422.
DOI:10.1016/j.colsurfb.2020.111422 |
| [21] |
Chadha A, Venkataramana S, Preetha R, et al. Candida parapsilosis: A versatile biocatalyst for organic oxidation-reduction reactions[J]. Bioorg Chem, 2016, 68: 187–213.
DOI:10.1016/j.bioorg.2016.08.007 |
| [22] |
Chan P H, Tsai S W. Two-step desymmetrization of dipyrazolidyl 3-phenylglutarate via lipasecatalyzed hydrolysis in organic solvents[J]. Chem Eng Sci, 2016, 139: 41–48.
DOI:10.1016/j.ces.2015.09.024 |
| [23] |
Yuan B, Debecker D P, Wu X F, et al. One-pot chemoenzymatic deracemisation of secondary alcohols employing variants of galactose oxidase and transfer hydrogenation[J]. ChemCatChem, 2020, 12(24): 6191–6195.
DOI:10.1002/cctc.202001191 |
| [24] |
Heath R S, Thompson M P, Turner N J, et al. Kinetic resolution and deracemization of racemic amines using a reductive aminase[J]. ChemCatChem, 2018, 10(3): 515–519.
DOI:10.1002/cctc.201701484 |
| [25] |
Pontini M, Hussain S, Turner N J, et al. Combined imine reductase and amine oxidase catalyzed deracemization of nitrogen heterocycles[J]. ChemCatChem, 2016, 8(1): 117–120.
DOI:10.1002/cctc.201500822 |
| [26] |
Schreier M R, Ward T R, Wenger O S, et al. Enantioselective synthesis of amines by combining photoredox and enzymatic catalysis in a cyclic reaction network[J]. Chem Sci, 2018, 9(22): 5052–5056.
DOI:10.1039/C8SC01561A |
| [27] |
Ferraz C A, Sergio G G, Dalmomico G, et al. Synthesis and characterization of a magnetic hybrid catalyst containing lipase and palladium and its application on the dynamic kinetic resolution of amines[J]. Mol Catal, 2020, 493: 111106.
DOI:10.1016/j.mcat.2020.111106 |
| [28] |
Magallanes-Noguera C, Ferrari M M, Orden A A, et al. Deracemization of secondary alcohols by chemo-enzymatic sequence with plant cells[J]. J Biotechnol, 2012, 160(3): 189–194.
|
| [29] |
Nosek V, Misek J. Chemoenzymatic deracemization of chiral sulfoxides[J]. Angew Chem Int Ed, 2018, 57(31): 9849–9852.
DOI:10.1002/anie.201805858 |
| [30] |
Yun I, Park J Y, Park J, et al. Base-free dynamic kinetic resolution of secondary alcohols with a ruthenium-lipase couple[J]. J Org Chem, 2019, 84(24): 16293–16298.
DOI:10.1021/acs.joc.9b02510 |
| [31] |
Hsiao H T, Lin S Y, Tsai S W. Quantitative insights and improvements of enzyme activity and stereoselectivity for CALB-catalyzed alcoholysis in two-step desymmetrization[J]. J Mol Catal BEnzym, 2016, 127: 82–88.
DOI:10.1016/j.molcatb.2016.02.013 |
| [32] |
Schober M, Valkov E, Heberlein M, et al. Structural and mechanistic analysis of the choline sulfatase from Sinorhizobium: A class I sulfatase specific for an alkyl sulfate ester[J]. J Mol Biol, 2018, 430(7): 1004–1023.
DOI:10.1016/j.jmb.2018.02.010 |
| [33] |
Wu Y W, Kong X D, Zhu Q Q, et al. Chemoenzymatic enantioconvergent hydrolysis of p-nitrostyrene oxide into (R)-p-nitrophenyl glycol by a newly cloned epoxide hydrolase VrEH2 from Vigna radiata[J]. Catal Commun, 2015, 58: 16–20.
DOI:10.1016/j.catcom.2014.08.020 |
| [34] |
Palombo E, Audran G, Monti H, et al. Enantioconvergent access to the enantiomerically pure building blocks (+)-or (-)-4-hydroxy-3-methyl-2-cyclohexenone using a chemoenzymatic process[J]. Synlett, 2006, 3: 403–406.
|
| [35] |
Sakai K, Fujii N, Chukeatirote E. Racemization of L-lactic acid in pH-swing open fermentation of kitchen refuse by selective proliferation of Lactobacillus plantarum[J]. J Biosci Bioeng, 2006, 102(3): 227–232.
DOI:10.1263/jbb.102.227 |
| [36] |
Xu J H, Yao L G, Kan Y C, et al. High level and enantioselective production of L-phenylglycine from racemic mandelic acid by engineered Escherichia coli using response surface methodology[J]. Enzyme Microb Tech, 2020, 136: 109513.
DOI:10.1016/j.enzmictec.2020.109513 |
| [37] |
Voss C V, Gruber C C, Kroutil W. Deracemisation of secondary alcohols via biocatalytic stereoinversion deracemisation of secondary alcohols via biocatalytic stereoinversion[J]. Synlett, 2010, 7: 991–998.
|
| [38] |
Zambrano G, Chino M, Renzi E, et al. Clickable artificial heme-peroxidases for the development of functional nanomaterials[J]. Biotechnol Appl Bio, 2020, 67(4): 549–562.
DOI:10.1002/bab.1969 |
| [39] |
Yuan Z S, Ke Z W, Qiu Y H, et al. Prewetting polypropylene-wood pulp fiber composite nonwoven fabric for oil-water separation[J]. ACS Appl Mater Inter, 2020, 12(41): 46923–46932.
DOI:10.1021/acsami.0c12612 |
| [40] |
Pagnout C, Sohm B, Razafitianamaharavo A, et al. Pleiotropic effects of rfa-gene mutations on Escherichia coli envelope properties[J]. Sci Rep, 2019, 9: 9696.
DOI:10.1038/s41598-019-46100-3 |
| [41] |
Chen L C, Yi Z L, Fang Y, et al. Uncovering key residues responsible for the thermostability of a thermophilic 1, 3(4)-β-d-glucanase from Nong flavor Daqu by rational design[J]. Enzyme Microb Tech, 2020, 142: 109672.
DOI:10.1016/j.enzmictec.2020.109672 |
| [42] |
Zhou P P, Xu N N, Yang Z F, et al. Directed evolution of the transcription factor Gal4 for development of an improved transcriptional regulation system in Saccharomyces cerevisiae[J]. Enzyme Microb Tech, 2020, 142: 109675.
DOI:10.1016/j.enzmictec.2020.109675 |
| [43] |
Markel U, Lanvers P, Sauer D F, et al. A photoclick-based high-throughput screening for the directed evolution of decarboxylase OleT[J]. Chem-Eur J, 2021, 27(3): 954–958.
DOI:10.1002/chem.202003637 |
| [44] |
Link A, Grathwol C W, Wu S K, et al. Directed evolution of a halide methyltransferase enables biocatalytic synthesis of diverse SAM analogues[J]. Angew Chem Int Ed, 2021, 60(3): 1524–1527.
DOI:10.1002/anie.202013871 |
| [45] |
Zhang Y Y, Zhao Y J, Li R, et al. Bioinspired NADH regeneration based on conjugated photocatalytic systems[J]. Sol Rrl, 2021, 5(2): 2000339.
DOI:10.1002/solr.202000339 |
| [46] |
Gu F J, Wang Y Z, Meng Z H, et al. A coupled photocatalytic/enzymatic system for sustainable conversion of CO2 to formate[J]. Catal Commun, 2020, 136: 105903.
DOI:10.1016/j.catcom.2019.105903 |
| [47] |
Preissler J, Reeve H A, Zhu T Z, et al. Dihydrogen-driven NADPH recycling in imine reduction and P450-catalyzed oxidations mediated by an engineered O2-tolerant hydrogenase[J]. ChemCatChem, 2020, 12(19): 4853–4861.
DOI:10.1002/cctc.202000763 |
 2021, Vol. 35
2021, Vol. 35


